안녕하세요. 휴먼스케이프에서 개발자로 일하고 있는 브루노입니다.
이 번 포스트에서는 annotation(VCF와 같은 변이 파일에 상세 정보를 첨가하는 작업) tool 중 하나인 Annovar를 간략히 소개하겠습니다.
Annovar란?
유전자 검사 후 분석 결과를 내기 까지는 아래와 같은 과정을 거칩니다.
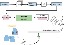
Annovar 동작 개요
유전체 분석을 위해서는,
시퀀싱 후에 각 read를 정리하여 BAM 파일을 추출하고
일련의 변이를 담은 genetic variants 파일을 만들어 냅니다 (ex. VCF)
만들어진 genetic variants 파일을 annotation tool(위 예에서 Annovar)에 입력해서 각 변이에 대한 분석 결과를 계산하여 분석 결과 파일을 만들어 냅니다
분석 결과 파일을 보고 목적에 따라서 Clinical report를 만들어내게 됩니다.
이 때 Annovar는 각 변이에 분석 결과를 첨가하는 annotation을 하는 소프트웨어, 즉 annotation tool입니다.
Annovar input file
Annovar는 자체 input 파일 형식을 가지고 있습니다. 각 필드가 탭으로 분리되어 있는 TSV(Tab Seperated Value) 텍스트 파일입니다. 공식 문서를 보면 space 5개도 가능하다고 하는데 아마도 TSV 형태가 일반적으로 쓰이는 것 같습니다.
기본 필드는 순서대로 염색체, start position, end position, Reference Allelel(레퍼런스 유전자), Alternative Allele(변이 유전자) 입니다. 그 뒤에 나오는 필드들은 추가 정보입니다.
아래는 Annovar input file의 예시입니다.

Annovar input file 예시
각 라인은 하나의 변이를 나타냅니다.
가장 보편적으로 쓰이는 VCF 파일을 Annovar를 이용해서 annotate하기 위해서는 Annovar input format으로 변경을 해야 합니다. 변경은 Annovar를 실행할 때
-format vcf4
옵션을 추가하면 자동으로 수행됩니다.
Annovar 사용법
Annovar는 오픈 소스이므로 input 파일을 준비했다면 바로 사용할 수 있습니다. 아래 내용은 공식 문서의 user guide를 참고했습니다.
http://annovar.openbioinformatics.org/en/latest/user-guide/download/에서 사용등록 후에 다운로드 받습니다.
다운로드 받은 파일의 압축을 해제하고, 압축 해제한 폴더로 이동합니다.
필요한 DB 들을 다운 받습니다. 아래는 DB 다운로드 명령어 예시입니다.
$ annotate_variation.pl -buildver hg19 -downdb -webfrom annovar refGene humandb/ $ annotate_variation.pl -buildver hg19 -downdb cytoBand humandb/ $ annotate_variation.pl -buildver hg19 -downdb -webfrom annovar exac03 humandb/ $ annotate_variation.pl -buildver hg19 -downdb -webfrom annovar avsnp147 humandb/ $ annotate_variation.pl -buildver hg19 -downdb -webfrom annovar dbnsfp30a humandb/
다운로드 받은 후, 아래 명령어를 이용해서 각 DB의 정보를 이용해 input의 gene들에 annotation을 진행합니다.
$ table_annovar.pl example/ex1.avinput humandb/ -buildver hg19 -out myanno -remove -protocol refGene,cytoBand,exac03,avsnp147,dbnsfp30a -operation gx,r,f,f,f -nastring . -csvout -polish -xref example/gene_xref.txt
Annovar 결과 파일
Annovar 결과 파일은 input 파일에 annotation 필드들이 추가된 형태를 띄게 됩니다. 어떤 DB를 추가하느냐에 따라서 필드는 추가될 수도, 줄어들 수도 있습니다. 필드명 또한 변경 가능합니다.
그렇기 때문에 Annovar로 annotation 된 결과 파일을 이용하기 위해서는 필드명과 필드 자료형을 고정하고 진행하는 것이 좋습니다.
정리
유전체 annotation tool 중 하나인 Annovar에 대해서 간략하게 소개했습니다. Annovar 외에도 SnpEff나 vep와 같은 많은 annotation tool이 있습니다. 대부분 유사한 방법으로 수행하므로 Annovar의 동작법을 잘 익히신다면 다른 annotation tool들을 익히는 데 많은 도움이 될 것입니다.
읽어 주셔서 감사합니다.
Annovar에 대한 더욱 자세한 내용은 공식 문서를 참고해주세요.
Get to know us better! Join our official channels below.
Telegram(EN) : t.me/Humanscape KakaoTalk(KR) : open.kakao.com/o/gqbUQEM Website : humanscape.io Medium : medium.com/humanscape-ico Facebook : www.facebook.com/humanscape Twitter : twitter.com/Humanscape_io Reddit : https://www.reddit.com/r/Humanscape_official Bitcointalk announcement : https://bit.ly/2rVsP4T Email : [email protected]