안녕하세요. 휴먼스케이프에서 개발자로 일하고 있는 브루노입니다.
이 번 포스트에서는 유전자 정보를 저장하는 양식 중 하나인 VCF에 대해서 설명 드리겠습니다.
VCF 형태
VCF는, 각 컬럼이 탭(tab)으로 구분되는 TSV (Tab-separated values) 텍스트 파일이며, UTF-8 인코딩을 사용합니다. 각 라인의 마지막은 LF(“ ”) 혹은 CR+LF(“ ”)으로 되어있습니다.
라인은 아래와 같은 세 종류의 라인이 존재합니다.
##으로 시작하는 메타 정보(meta-information) 라인
#으로 시작하는 한 줄의 헤더 라인
유전자 단위의 변이 별로 한 줄을 이루는 데이터 라인

VCF 예시
VCF 파일을 텍스트 에디터로 열어보면 위 순서대로 적힌 라인들을 볼 수 있습니다.
이제 각 라인 유형별로 설명을 시작하겠습니다.
Meta-information lines
VCF에서 맨 위에서 볼 수 있는, ##으로 시작하는 라인들이 바로 Meta-information line입니다. 파일 전체의 메타 데이터를 담고 있습니다.
Meta-information lines의 형태
Meta-information key=value 쌍으로 이루어져 있으며, value는 캐럿(“<>”)으로 감싸져있습니다.
meta-information은 File Format 라인을 제외하고는 없어도 무방하지만, VCF의 정보를 파싱하기 위해서, 그리고 VCF의 binary 형식인 BCF로의 변환을 위해서는 필요합니다.
그럼 meta-information line의 몇 가지 예시를 들어 설명 드리겠습니다.
File Format
file format은 meta-information line 중 항상 제일 먼저 나와야 합니다. 아래와 같이 VCF 파일의 버전을 나타냅니다.
##fileformat=VCFv4.3
Information field format
Information field format 라인은 아래와 같이 ##INFO 로 시작하고, “<>”로 감싸진 값을 가집니다.
후에 설명할 data line에는 각 변이의 추가 정보를 제공하는 필드인 INFO 필드가있습니다. meta-information line 중, Information field format은 그 데이터 line의 INFO 필드의 format을 알려주는 라인입니다.

Information field format 라인 예시
Filter field format
filter field format 라인은 ##FILTER로 시작합니다.
데이터 라인에서 변이 검사 퀄리티를 나타내는 FILTER 필드의 format을 알려 줍니다.
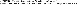
Filter field format 라인 예시
Individual format field format
Individual format field format 라인은 ##FORMAT 으로 시작합니다.
데이터 라인에서 genotype 별 데이터가 나오는데, 이 때 여기에 어떤 데이터가 어떤 format으로 들어가 있는지 알려줍니다.
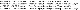
Individual format field format 라인 예시
이 외에도 여러 종류의 meta-information lines 가 존재하지만, 주로 위에 설명한 라인들이 중요한 정보를 담고 있습니다. 더 다양한 정보는 공식 문서를 확인해주세요.
Header line
헤더 라인은, 데이터 라인에 어떤 필드들이 있는지 알려주는 라인입니다.

Header line 예시
데이터 라인에서 헤더 라인에 정의된 순서대로 한 필드에 해당 데이터가 나열됩니다.
만약 genotype 데이터가 있는 VCF라면, FORMAT 필드와 sample ID가 헤더 라인에 추가됩니다.
Data lines
모든 실제 데이터들은 데이터 라인에 있습니다. meta-information과 header line에 정의된 format에 맞춰서, 한 줄에 한 변이에 대한 정보들을 담고 있습니다.
만약 특정 라인에서 필드에 해당하는 데이터가 없을시, ‘.’ 이 대신 입력됩니다.

data lines 예시
데이터 라인의 각 필드에 대해 설명해보겠습니다.
CHROM
chromosome: 변이가 위치하는 염색체 번호를 나타냅니다.
POS
position: 변이의 염색체 내 위치를 나타냅니다.
ID
identifier: 유전자 ID. 일반적으로 dbSNP 내에서의 유전자 ID를 사용합니다. 만약 ID를 찾을 수 없는 경우 ‘.’이 입력됩니다.
REF
reference base(s): 해당 변이의 위치에서 정상이라고 판단되는 원래 base 시퀀스를 나타냅니다. 길이 1 이상의 정상 bases가 나열됩니다. bases이므로 A, C, G, T, N로 이루어진 문자열이 나옵니다.
ALT
alternate base(s): 유전자 검사에서 나온, reference bases와 다른 유전자, 즉 변이 bases를 나타냅니다
QUAL
quality: Phred 품질 점수를 나타냅니다. 유전 검사의 신뢰도를 나타내는 수치입니다.
FILTER
filter status: 품질 점수에 따라서 신뢰할 수 있는지를 나타냅니다. 특정 신뢰도(filter)를 만족하는 경우 ‘PASS’로 표시되고, 특정 신뢰도를 만족하지 못하는 경우 “q10;s50”와 같이 만족하지 못한 filter에 대한 정보가 표시됩니다. 만약 filter가 적용되지 않은 경우 ‘.’으로 표시됩니다.
INFO
additional information: 변이에 대한 추가 정보를 담고 있는 필드입니다.
세미콜론(;)으로 구분된 여러 데이터가 key=value[,value] 형태로 나열된 문자열입니다. 각 필드에 대한 설명은 meta-information에 정의됩니다.
FORMAT
genotype fields format: 뒤에 나오는 genotype fields에 어떤 순서대로 데이터가 나올지 형태를 알려주는 필드입니다. 콜론(:)으로 구분된 테이터 key들이 나열됩니다.
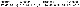
format과 genotype 필드 예시
위에 예시를 보면, FORMAT에서 뒤에 genotype fields에 GT, GQ, DP, HQ 순서대로 콜론(:)으로 구분된 데이터들이 나올 것이라는 걸 나타냅니다.
각 key들이 어떤 의미인지는 meta-information의 FORMAT 라인에서 알려줍니다.
Genotype fields
샘플 별로 변이에 대한 추가 정보를 제공합니다. 앞의 FORMAT 필드에서 표시한대로 각 데이터를 콜론(:)으로 구분해서 나열합니다.
위의 FORMAT필드에서의 예시를 보면, NA0001의 샘플에서는 GT:0|0, GQ:48, DP:1, HQ:51,51임을 나타냅니다.
정리
유전체 데이터를 다루는 보편적인 형식인 VCF 파일의 형태에 대해서 간략하게 소개했습니다.
읽어 주셔서 감사합니다.
더 자세한 설명은 공식 문서 The Variant Call Format Specification를 확인해주세요.
Get to know us better! Join our official channels below.
Telegram(EN) : t.me/Humanscape KakaoTalk(KR) : open.kakao.com/o/gqbUQEM Website : humanscape.io Medium : medium.com/humanscape-ico Facebook : www.facebook.com/humanscape Twitter : twitter.com/Humanscape_io Reddit : https://www.reddit.com/r/Humanscape_official Bitcointalk announcement : https://bit.ly/2rVsP4T Email : [email protected]